肠道微生物竞争有限资源,加速抗生素的发现
人类的肠道内平均含有大约100万亿个细菌,这些微生物为了生存而竞争有限的资源。宾夕法尼亚大学工程与应用科学学院生物工程系助理教授César de la Fuente说:“这是一个非常激烈的环境。所有这些细菌共存的同时也在互相竞争。而这种环境恰恰能够激发微生物产生新的机制。”
在这种冲突中,de la Fuente实验室看到了新抗生素的潜力,如果肠道内的细菌为了生存必须发展出新的策略,那么为什么不利用这些策略来对抗它们呢?
在一项新的研究中,de la Fuente实验室和斯坦福大学的Ami S.Bhatt教授对近2000人的肠道微生物组(gut microbiome)进行了调查,发现了数十种潜在的新抗生素。相关研究结果于2024年8月19日在线发表在Cell期刊上,论文标题为“Mining human microbiomes reveals an untapped source of peptide antibiotics”。
de la Fuente认为生物学是一种信息源。“一切都是代码,”他说,“如果我们能够开发出一种算法来解读这些代码,就能极大地加速抗生素的发现。”近年来,de la Fuente实验室因其在各种意想不到的地方发现候选抗生素而受到关注,包括已灭绝生物如尼安德特人和长毛象的遗传信息,以及利用人工智能分析大量细菌遗传物质的工作。
他们的目标之一是分析全球生物信息,作为抗生素和其他有用分子的来源。他们不再依赖传统的采集土壤或水样并从中提取活性化合物的方法,而是利用基因组、宏基因组和蛋白质组中的大数据。这使得他们能够以更快的速度发现新的抗生素。
考虑到细菌的快速进化,de la Fuente和他的合著者假设,一个充满竞争的环境,例如人类的肠道,可能是无数未被发现的抗菌化合物的宝库。“当资源稀缺时,生物学才会真正展现出其创新的一面,”de la Fuente说。
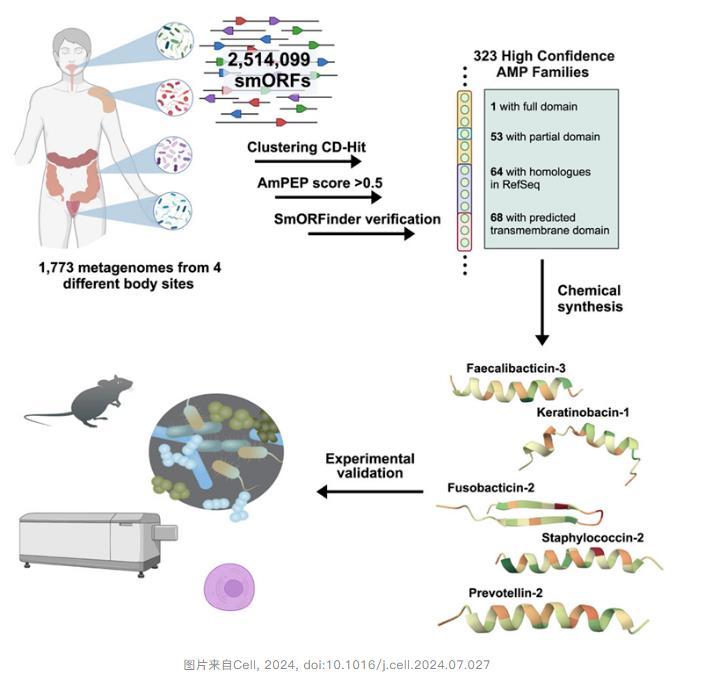
研究团队专注于肽,即氨基酸短链,这类物质之前被证明有潜力成为新的抗生素。他们通过计算分析了超过40万种蛋白质,利用人工智能读取遗传密码,并通过训练来预测哪些遗传序列可能具有抗菌特性。
论文的第一作者,de la Fuente实验室的助理研究员Marcelo D.T.Torres指出,“这些分子的构成与传统意义上的抗生素不同。我们发现的这些化合物构成了一个新的类别,它们的独特性质将有助于我们理解和扩展抗菌剂的序列空间。”
当然,这些预测需要通过实验来验证。在筛选出数百种候选抗生素后,研究团队选择了78种进行实际细菌测试。在合成这些多肽之后,他们让细菌培养物接触到每种多肽,并观察哪种肽能成功抑制细菌生长。此外,他们还在动物模型中测试了这些候选抗生素。
结果表明超过一半的肽有效,即它们抑制了有益或有害细菌的生长,而主要候选抗生素prevotellin-2的抗感染能力与FDA批准的抗生素多粘菌素B相当,显示出人类肠道微生物组可能蕴藏着未来可用于临床治疗的抗生素。
Bhatt评论道,“prevotellin-2的活性与我们最后防线之一的多粘菌素B相当,这个发现让我感到十分惊讶。这表明,从人类微生物组中挖掘新型抗菌肽,对于科学家、医生以及患者来说,是一条极具前景的道路。”
相关新闻推荐
1、临床结核分枝杆菌体外药敏试验:分子DST检测VS表型DST药敏诊断技术(二)
